NGS捕获探针应用与设计难点
在NGS全测序中,数据量庞大,数据分析困难。针对某些疾病或肿瘤相关的目的基因进行测序时,通常选择靶向捕获目标区域进行实验。这种方法测序成本更低,测序深度更高,数据有效率也更高,适用于检测SNP、InDel、CNV、Fusion等突变类型。
靶向捕获主要有两种方式:
1.多重PCR:
通用扩增子引物扩增的方式得到的基因片段,能有效富集目的片段,但单次实验的扩增子数量受限、设计难度高。
2.杂交捕获:
生物素修饰的探针通过碱基互补配对去捕获目的基因片段。当探针和目标区段杂交完成后,通过磁珠将探针吸附使得目的片段和非目标片段分开,对获得目的片段进行测序。相较于多重PCR,杂交捕获可检测变异类型多、大小panel都适用、探针对错配的容忍度高、检测范围广。
百力格捕获探针设计
为了解决杂交捕获探针设计中面临的捕获特异性和均一性问题,百力格NGS团队结合经验,自主研发杂交捕获探针设计工具 Smartbaits,能够方便、高效、高质量的完成探针设计。
设计优势
2、优化的算法:综合考虑了探针的GC 平衡、特异性、探针之间的同源和异源二聚体,发卡结构等。使用更细的颗粒度寻找最佳探针方案,同时确保使用更少的探针达到覆盖度最大化,减少成本。
3、更加灵活的参数选择:可选择1x,2x tiling和overlap等不同的密度模式;可以选择不同的参考基因组或者目标序列,不仅限于常见模式生物或者人类基因组;根据序列特异性,极端GC以及二级结构区域对探针风险进行分级等。
4、灵活的设计方案:不同区间采用不同的方案。基因区间和间区、SNP骨架、MSI采用不同的优化算法进行设计。
5、可持续性:通过spike-in 的方式,可以在原有的panel中另外添加探针形成新的panel
6、使用灵活、门槛低:smartbaits采用SQL关系型数据库后端,以最小化需要高内存负载和大量I/O计算时间的数据文件问题。这使得用户可以快速使用多个不同的筛选标准重新分析,并最小化使用门槛。
设计原理
对于不同区域设计采用不同的方式设计。
1.普通区域设计:以中心对称原则,在滑窗可移动范围内寻找最佳探针集合。
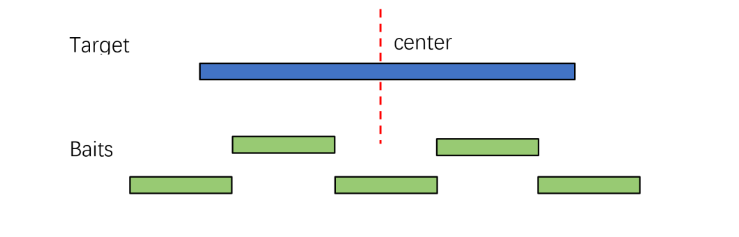
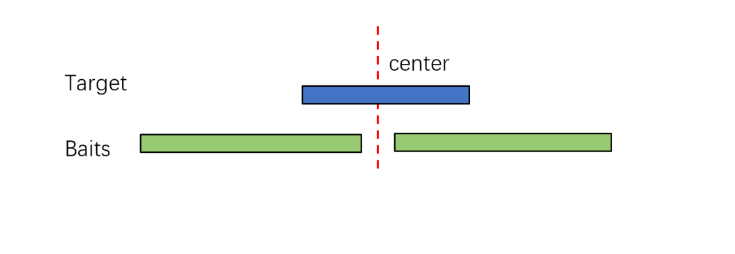
2.微卫星序列设计:大多微卫星序列较短,重复序列高,常规设计通常不能达到理想的捕获结果,因此采用优化的双端探针分别覆盖目标区域的两端的策略。
设计流程

输入质检
对于用户输入的内容转化为fasta序列进行质检分析,主要包括序列GC含量、Tm、序列复杂度分析等,将分析结果保存至数据库中。
1、输入
接受用户输入.bed文件格式、基因组.fasta或者gene symble 名称格式作为输入文件。普通设计为1x tiling,用户可以根据需要调整为2x tiling。程序会将输入的信息进行分析并保存在数据库当中。
2、输入质检
对于用户输入的内容转化为fasta序列进行质检分析,主要包括序列GC含量、Tm、序列复杂度分析等,将分析结果保存至数据库中。
3、设计探针
应用滑窗对候选目标区域进行分割,以探针长度为120 bp生成所有可能的探针,采用中心对称原则从左到右进行以step=1的步长生成候选探针集合。
4、探针筛选
分析所有的探针集合并质检。对每一个探针使用blastn 进行特异度分析,使用用户设置的参数通过决策树筛选探针集。主要决策点包括序列特异度、GC、Tm、二级结构等,得到最终探针集。
5、设计报告
设计完毕后会对探针进行风险分级:green 代表安全探针,保证捕获效率和特异性,yellow 代表有一定的特异度风险,black 代表特异性或者捕获效率较差,不建议添加。
最终生成三份结果:
1. 探针设计报告
含有探针覆盖度,探针数量等信息的PDF报告;
2. 通过探针信息(pass baits)
包括所有合格的探针信息,内含每一个探针的质检数据;
3. 所有探针信息(all baits)
含有所有的探针信息和质检数据,包括black、yellow探针。
性能对比
下表是使用百力格技术smartbaits和Ixx公司技术分别对基因KRAS、ALK、ROS1、EGFR、BRAF(CDS区)设计探针后进行杂交捕获实验结果对比。在downsample 到同等数据量下,百力格获得了更高的捕获效率,更高的平均测序深度,更低的fold80,并且达到了100%的 1x 覆盖度。

应用范围
使用杂交捕获的靶向高通量测序技术可以应用于多种类型的研究,例如外显子组捕获,单核苷酸多态性(SNP),遗传性肿瘤检测,复杂疾病诊断,目的片段个性化捕获分析等。
使用方法
您可以通过邮件blg-mkt@bioligo.com、公众号私信或当地销售联系我们进行设计,Smartbaits未来也将在百力格官网上线,敬请期待!








