在讲解精准定量的计算之前,我们需要了解一下分光光度法的原理,由于Oligo中的碱基之间存在共轭双键,共轭双键对光具有强吸收的特点,因此当测量吸收光谱时,在波长260 nm或接近260 nm处都有一个吸光度(Absorbance, A260),如下图所示:
但是,我们在引物管上通常见到的是OD值,什么是OD₂₆₀?
OD₂₆₀是指在260 nm波长处的光密度(Optical Density, OD),OD260测量单位定义为使用1cm的光程(pathlength)、测量260 nm波长处、1 ml寡核苷酸溶液产生读数为1.0时所需要的寡核苷酸浓度。OD₂₆₀值与A₂₆₀值相关,计算公式为:
OD₂₆₀ = A₂₆₀ x 体积(ml) /光程(cm)
由于OD260测量时体积为1 ml,光程为1 cm,所以OD₂₆₀值= A₂₆₀值。
1. 常见定量误区
DNA定量和RNA定量一般称为核酸定量,通常测量样品中DNA或RNA的平均浓度,然后再进行下游实验。通常,在260 nm处的吸光度即可用于测量溶液中存在的核酸的浓度。近似的转换系数为:
a. 双链DNA(ds DNA)约为50 μg/OD260;
b. 单链DNA(ss DNA)约为33 μg/OD260;
c. 单链RNA约为40 μg/OD260
当OD=1时,依据:
摩尔数(μmol)=质量/分子量=(转换系数x OD)/分子量(MW)
代入浓度计算公式:
摩尔浓度(μmol/L,μM) =(摩尔数/体积)x 1000
*分子量(MW)的计算方式见文章最后部分
值得注意的是,转换系数计算公式只适用于长的、碱基随机排列的序列,对于长度较短的序列,重复序列,以及带有特殊修饰基团的Oligo序列,每个碱基的组成、排列顺序和修饰都会影响序列计算,导致定量准确率较低。
2. 什么是Oligo精准定量?
对于Oligo的定量,为了保证得到准确的定量结果,通常不能直接使用仪器测定的ng/μl数值,尤其对于带有修饰的Oligo探针, 直接使用仪器测定的ng/μl数值会使定量结果出现很大的偏差。这时就需要引入对于寡核苷酸的定量计算至关重要的数值:消光系数(Extinction coefficient,ε)。
消光系数和最大吸收波长都会随着共轭双键数量的增加而增加,每个碱基的吸光度不同,碱基的组成和排列顺序都会影响吸光度,因此在定量过程中引入了消光系数进行计算。消光系数依赖于精确的核苷酸组成和排列顺序,对于每个寡核苷酸来说消光系数是唯一的,因此,通过计算每个寡核苷酸的消光系数精确值,可以获得最高的定量准确度。
3. 如何计算Oligo的消光系数(ε)?
百力格使用邻近法(Nearest neighbor method)计算每个合成的寡核苷酸的消光系数,然后用来计算每个寡核苷酸的产量。计算公式为:
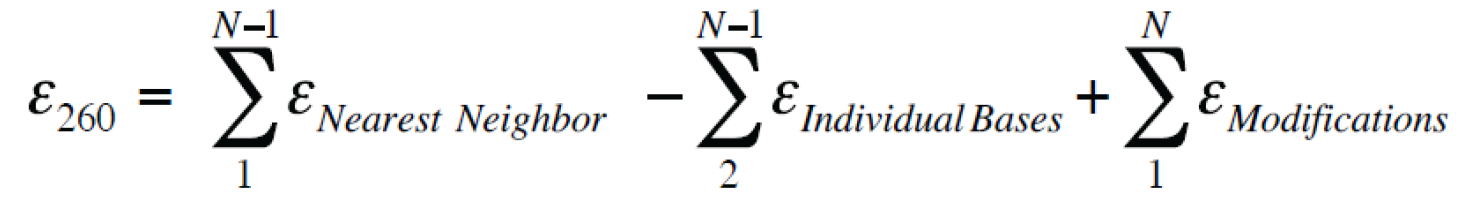
其中:ε260:寡核苷酸序列在 260 nm 处的消光系数,单位为(L/mol·cm);
Σ1N-1εNearest Neighbor:相邻碱基对的消光系数之和;
Σ2N-1εIndividual Bases:单个碱基的消光系数之和;
Σ1NεModifications:修饰的消光系数之和。
参考邻近值:
5'/3' position | Adenine | Guanine | Cytosine | Thymine | Uracil |
Adenine | 27,400 | 25,000 | 21,200 | 22,800 | 24,600 |
Guanine | 25,200 | 21,600 | 17,600 | 20,000 | 20,000 |
Cytosine | 21,200 | 18,000 | 14,600 | 15,200 | 17,200 |
Thymine | 23,400 | 19,000 | 16,200 | 16,800 | N/A |
Uracil | 24,000 | 21,200 | 16,200 | N/A | 19,600 |
单个碱基值:
Adenine | Guanine | Cytosine | Thymine | Uracil |
15,400 | 11,500 | 7,400 | 8,700 | 9,900 |
*即使邻近法相对准确,但是其结果也可能与真值存在偏差。
4. 如何通过消光系数(ε)计算Oligo的浓度?
根据朗伯比尔定律(Beer-Lambert Law):
A₂₆₀ = ε₂₆₀ × C × p (1)
推导出:
C = A₂₆₀ /(ε₂₆₀ × p) (2)
其中:c:Oligo浓度(mol/L);A:吸光度;ε:消光系数(L/mol·cm); p:光程(cm)
当光程p=1 cm,进行单位换算后,
可得百力格浓度计算公式:
C = A₂₆₀ × (1/ε₂₆₀) × 106= A₂₆₀ × nmol/OD
其中:c:Oligo浓度(μmol/L,μM);A:吸光度;nmol:物质的量单位;OD:光密度
5. 如何计算Oligo的分子量(MW)?
引物合成单上出现的另一个重要数据是分子量,MW用来将OD260单位转化为质量单位,
寡核苷酸的总分子量即所有碱基的原子量之和,再根据不同类型进行调整:
a. 无5’单磷酸盐的DNA序列:ssDNA-61.96 Da,dsDNA-123.38 Da;
b. 含5’单磷酸盐的DNA序列:ssDNA+17.04 Da,dsDNA+34.08 Da;
c. 含5’三磷酸盐的RNA序列:RNA+ 159.0 Da;
单位为道尔顿(Dalton,Da),1 Da ≈ 1 g/mol
Nucleotide | ss DNA | ds DNA | RNA |
Adenine | 313.21 Da | 616.78 Da | 329.21 Da |
Guanine | 329.21 Da | 617.88 Da | 345.21 Da |
Cytosine | 289.18 Da | 617.88 Da | 305.18 Da |
Thymine | 304.20 Da | 616.78 Da | N/A |
Uracil | N/A | N/A | 306.20 Da |
举个例子来看一下依据经验值的模糊浓度计算和百力格精确浓度计算之间的差别:
如ss DNA寡核苷酸序列5’-ATGGCTAC-3’
常规计算:
分子量MW=313.21x2+304.20x2+329.21x2+289.18x2-61.96=2409.64 Da≈2409.64 g/mol
摩尔浓度(μmol/L,μM)=(转换系数/分子量(MW))×1000
摩尔浓度(μmol/L,μM)=(33/2409.64)× 1000≈13.69 μM
百力格计算:
消光系数计算ε260=77,600 L/mol·cm
C (μmol/L,μM)= A260×(1/ε260)×106=A260×nmol/OD=A260 ×106/77,600≈12.89 μM
*1 μM=1 μmol/L=1 pmol/μl
百力格Oligo合成
相较于仪器测定的ng/μl数值,百力格的浓度计算方式更为准确,尤其是对于长度较短的或者带有特殊修饰基团的Oligo序列,更加贴近于真值。
百力格生物依托专业的技术人员、自动分装设备和成熟的合成纯化工艺,尽可能减少操作过程中的损失,确保Oligo精准和系统性定量
参考文献:
[1]https://sg.idtdna.com/pages/education/decoded/article/oligo-quantification-getting-it-right
[4]Cavaluzzi MJ, Borer PN. (2004) Revised UV extinction coefficients for nucleoside–5‘–mono¬phosphates and unpaired DNA and RNA. Nucleic Acids Res, 32(1) e13.
[5]Cantor CR, Warshaw MM, Shipiro H. (1970) Oligonucleotide interactions. III. Circular dichroism studies of the conformation of deoxyoligonucleolides. Biopolymers, 9(9):1059−1077.








